
INTRODUCCIÓN A LA GENÉTICA MOLECULAR


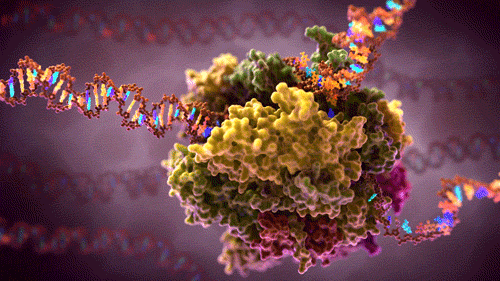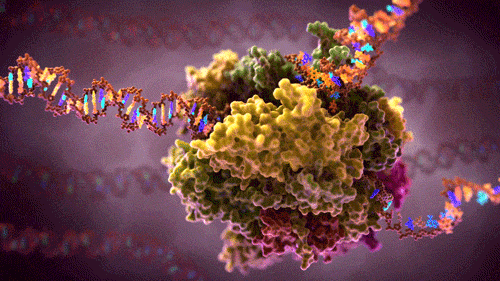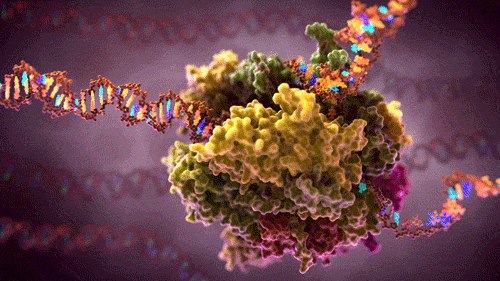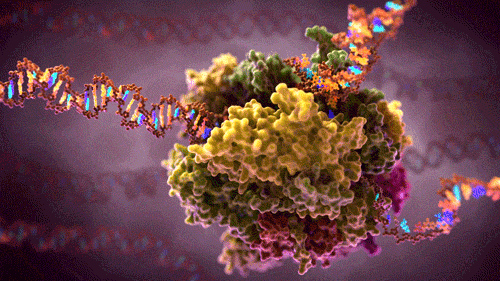
La genética molecular estudia cómo la información genética codificada en el ADN se transfiere y se expresa dentro de la célula. Esta información fluye mediante dos grandes procesos:
-
Replicación del ADN: ocurre en la fase S del ciclo celular, permitiendo la transmisión de la información genética durante la mitosis (de célula a célula) y la reproducción (de individuo a individuo).
-
Expresión génica:
-
Transcripción (síntesis de ARN mensajero, ARNm)
-
Traducción (síntesis de proteínas a partir del ARNm).

El Dogma Central de la Biología Molecular
Francis Crick (1970) propuso que la información genética fluye de:
-
ADN → ARN → Proteína
Este dogma se amplió con descubrimientos posteriores:
-
Transcripción inversa: virus como el VIH usan la enzima transcriptasa inversa para copiar su ARN en ADN.
-
Replicación de ARN: llevada a cabo por enzimas replicasas en virus de ARN.

Flujo de Información Genética en Células Eucariotas
-
Replicación y transcripción: ocurren en el núcleo.
-
Traducción: tiene lugar en los ribosomas del citoplasma.
Cada etapa depende de enzimas específicas que aseguran precisión y control.




Aporte de Rosalind Franklin
-
Obtuvo imágenes del ADN mediante difracción de rayos X, lo que permitió deducir su estructura helicoidal. Su trabajo fue crucial para que Watson y Crick propusieran la doble hélice.

Proceso mediante el cual se forman dos copias idénticas de la molécula original de ADN.
Enzimas implicadas:
-
ADN polimerasas (ADN pol): catalizan la unión de nucleótidos y también actúan como exonucleasas.
-
ARN polimerasa (ARN pol): sintetiza fragmentos cortos de ARN (cebadores).
-
Topoisomerasas / girasas: reducen la torsión del ADN.
-
Ligasas: sellan los espacios entre fragmentos de ADN.
En Procariotas:
-
La horquilla de replicación se forma desde un origen único.
-
Helicasas: separan las cadenas.
-
Proteínas SSB: estabilizan las cadenas abiertas.
Problemas resueltos:
-
La ADN pol no puede iniciar síntesis sin un cebador.
-
Solo sintetiza en dirección 5’ → 3’.
Cadena conductora:
-
ARN pol sintetiza el cebador.
-
ADN pol III extiende la cadena.
-
ADN pol I reemplaza el cebador por ADN.
-
Ligasa une los fragmentos.
Cadena retardada:
-
Primasa sintetiza varios cebadores.
-
ADN pol III forma los fragmentos de Okazaki.
-
ADN pol I reemplaza los cebadores.
-
Ligasa une los fragmentos.
Aporte de Francis Crick y James Watson
Propusieron el modelo de replicación semiconservativa: cada cadena original sirve de molde para una nueva.


Replicación del ADN


Transcripción
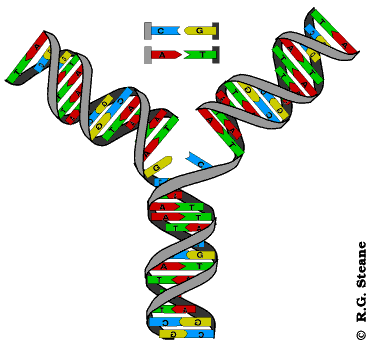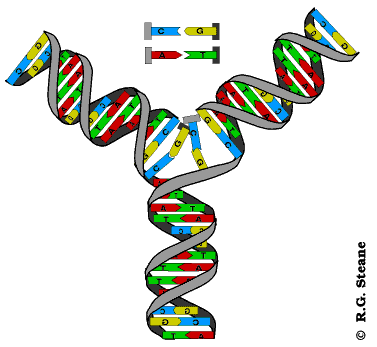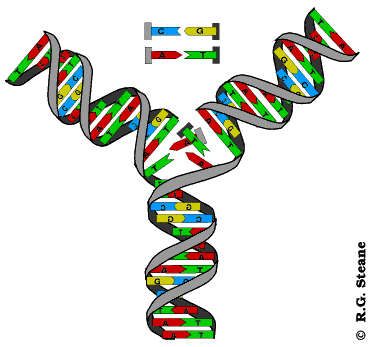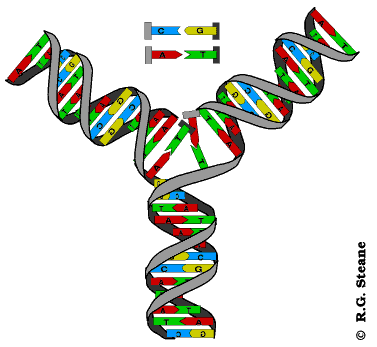
Proceso por el cual se sintetiza ARN a partir de ADN, produciendo una cadena complementaria.
Enzima principal: ARN polimerasa
Cataliza:
-
Separación de las cadenas de ADN.
-
Unión de ribonucleótidos en sentido 5’ → 3’ (sin cebador).
En Procariotas:
-
Inicio:
-
ARN pol reconoce promotores (posiciones –35 y –10) mediante la subunidad sigma.
-
Se posiciona en el nucleótido +1 e inicia la transcripción.
-
-
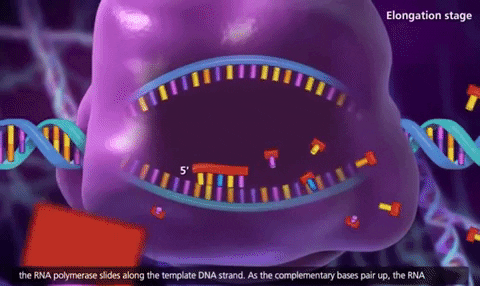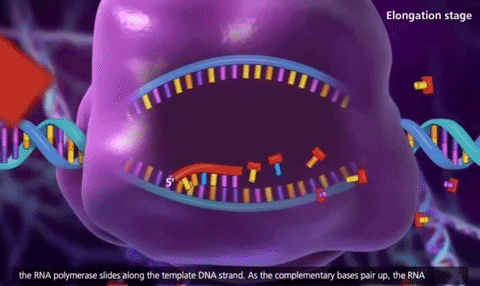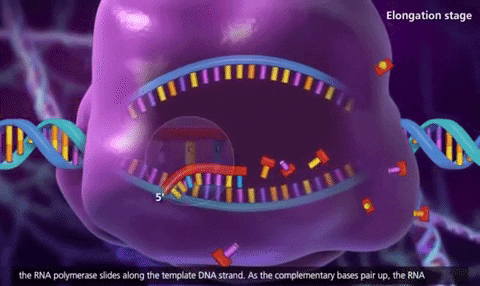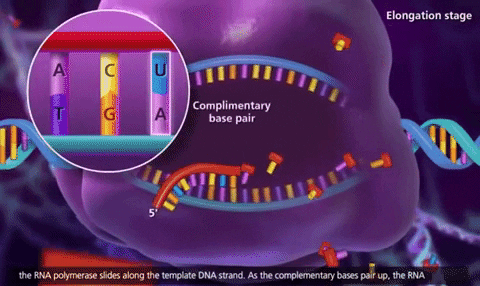
Elongación:
-
La ARN pol avanza añadiendo ribonucleótidos.
-
Se forma una burbuja transcripcional donde el ARN está temporalmente unido al ADN.
-
-
Terminación:
-
Estructuras tipo horquilla (bucles) causan el cese de la transcripción.
-
En Eucariotas:
-
Inicio:
-
Promotores ubicados cerca de la caja TATA (posición –25).
-
Factores de transcripción (TFs) permiten que la ARN pol II se una al ADN.
-
-
Elongación:
-
ARN pol II recorre la cadena molde 3’ → 5’.
-
Se sintetiza ARN en dirección 5’ → 3’, usando uracilo (U) en lugar de timina (T).
-
-
Terminación:
-
El ARN resultante es un transcrito primario que sufrirá modificaciones post-transcripcionales (capuchón, cola poli-A, corte y empalme).
-



Traducción
_edited.png)
Proceso mediante el cual se sintetiza una proteína a partir del ARNm.
Requisitos:
-
ARNm maduro
-
Ribosomas completos
-
ARNt cargados con aminoácidos
Código Genético:
-
Combinaciones de 3 nucleótidos (tripletes) llamados codones codifican 20 aminoácidos.
-
Es universal y degenerado (varios codones por aminoácido).
-
Codones especiales:
-
AUG: inicio (metionina)
-
UAA, UGA, UAG: terminación
-
Fases del proceso:
-
Activación de aminoácidos:
-
Enzimas aminoacil-ARNt sintetasas cargan aminoácidos en ARNt específicos según el anticodón.
-
-
Iniciación:
-
El ARNt con metionina (anticodón UAC) se une al codón de inicio AUG.
-
La subunidad mayor del ribosoma se une para formar el complejo de iniciación.
-
-
Elongación:
-
Dos sitios activos:
-
Sitio P: contiene el ARNt con el péptido.
-
Sitio A: entra el nuevo ARNt.
-
-
Se forma un enlace peptídico entre aminoácidos.
-
El ribosoma se desplaza codón por codón.
-
-
Terminación:
-
Al llegar a un codón de parada, se liberan:
-
La proteína
-
El ARNm
-
Las subunidades ribosómicas
-
-

Repasemos lo aprendido

Presione aquí para repasar lo aprendido......
Animación de ADN a ARN a proteína



